Estudio muestra cómo RAS normal interactúa con RAS mutado en células vivas
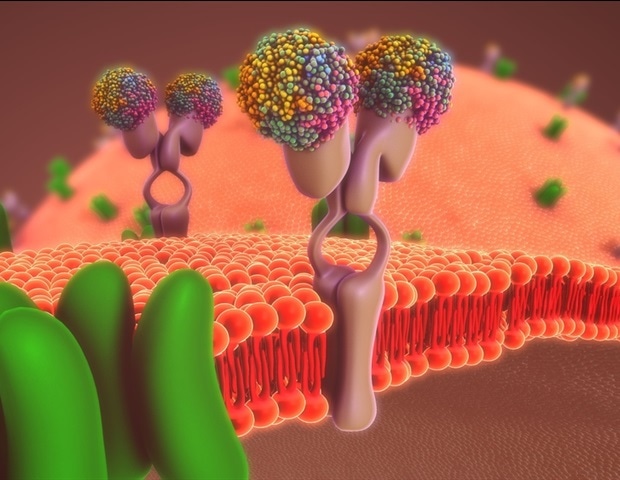
Muchos medicamentos contra el cáncer no logran atacar eficazmente los genes de cáncer mutados más comúnmente en humanos, llamados RAS. Ahora, el profesor de Salk Geoffrey Wahl y un equipo de científicos han descubierto detalles sobre cómo RAS normal interactúa con RAS mutado y otras proteínas en las células vivas por primera vez. Los hallazgos, publicados en The Proceedings of the National Academy of Sciences el 18 de mayo de 2020, podrían ayudar en el desarrollo de mejores terapias contra el cáncer dirigidas a RAS.
"Las proteínas RAS se han estudiado durante décadas porque el gen RAS está cambiado (mutado) en muchos tipos de cáncer, pero aún hay cosas nuevas que aprender a medida que desarrollamos herramientas más sofisticadas para estudiar el problema", dice Wahl, coautor correspondiente. , profesor en el Laboratorio de Expresión Genética de Salk y titular de la Cátedra Daniel y Martina Lewis. "Hemos identificado un nuevo mecanismo para regular la actividad de la enzima RAS que ayudará a informar estrategias terapéuticas para inhibir las proteínas RAS mutadas involucradas en el cáncer". La familia de genes RAS ayuda a regular la comunicación celular ("señalización") y el crecimiento. Sin embargo, investigaciones anteriores sugieren que el RAS mutado se desvía del RAS normal en su capacidad para regular los procesos que impulsan el crecimiento tumoral a través de múltiples tipos de cáncer, incluido el La mayoría de los cánceres pancreáticos. Los científicos han intentado durante mucho tiempo apuntar a la actividad de RAS relacionada con el cáncer, pero esto ha resultado muy difícil. Los esfuerzos para comprender qué proteínas RAS normales y mutantes interactúan dentro de la célula también han dado respuestas contradictorias debido a la dificultad de replicar el entorno celular en un tubo de ensayo.
Y aunque estudios anteriores han sugerido que las proteínas RAS normales pueden unirse a proteínas RAS mutadas para suprimir el crecimiento tumoral, se desconocía exactamente cómo ocurrieron estas interacciones. Mejoramos una tecnología genética existente desarrollada por nuestro laboratorio, que nos permite estudiar las interacciones de proteínas RAS instantáneamente en células vivas, la clave para comprender la función de RAS es poder analizar con precisión las interacciones de proteínas en la membrana celular. Esta nueva tecnología nos permite hacer eso.
" Yao-Cheng (Leo) Li, líder del estudio y científico del proyecto Salk, Instituto Salk Al igual que ver a un equipo de fútbol ejecutar sin esfuerzo una jugada complicada, el equipo utilizó su herramienta genética de alta potencia (que permite que las proteínas que interactúan se iluminen, como las luciérnagas) para examinar cómo interactúa RAS con otras proteínas, así como con su forma mutada, dentro de las células vivas. Descubrieron que se requería una proximidad cercana en la membrana celular para que una proteína RAS interactuara con otras proteínas RAS, comportamiento que el equipo acuñó "interacciones facilitadas por asociación de membrana" (MAFI). La membrana celular es necesaria para las interacciones RAS consigo misma y con algunas otras proteínas que se localizan en el mismo lugar en la membrana celular, por lo que tales interacciones no se encontraron previamente en estudios de probeta.
El equipo también descubrió inesperadamente un nuevo mecanismo para regular la cantidad de proteínas RAS en la célula. Descubrieron que si colocaban un pequeño fragmento de una proteína que interactúa fuertemente con RAS en la membrana, MAFI permitiría que esta proteína se una muy fuertemente a RAS, y esto podría inhibir mejor la función de RAS, creando un complejo RAS inactivo. La célula tiene un mecanismo para detectar y eliminar complejos RAS inactivos utilizando pequeñas estructuras llamadas lisosomas para realizar este "lavado de la casa". Debido a que la célula murió como resultado de la eliminación de las proteínas RAS, este hallazgo nuevo e inesperado puede ayudar en el desarrollo de nuevas terapias contra el cáncer. "Estos hallazgos definen nuevos mecanismos de regulación de señalización RAS", dice Nikki Lytle, autora del artículo y becaria postdoctoral de Salk. "Esto proporciona un modelo inesperado para la supresión de RAS, que podría conducir a nuevas estrategias para apuntar a RAS mutado en el futuro". En el futuro, los investigadores esperan que su descubrimiento se pueda utilizar para desarrollar una nueva clase de terapias dirigidas a RAS, que pueden requerir la administración de medicamentos a través de enfoques de vanguardia que involucran nanopartículas o virus que pueden atacar las células malignas.
Yorumlar
Yorum Gönder